Protein dizisi hizalaması, protein dizilerini karşılaştırarak benzerlikleri ortaya çıkarmak için temel bir yöntemdir ve modern biyoloji ile tıpta büyük önem taşımaktadır. Bu süreç, gen fonksiyonlarını aydınlatarak evrimsel ilişkileri yeniden yapılandırmaya yardımcı olur. Bu teknik, homoloji çıkarımı olarak adlandırılır ve ilaç geliştirme süreçlerine önemli katkılar sağlar. Bilim insanları yeni bir protein keşfettiklerinde veya tasarladıklarında, bunu bilinen protein dizileri ile hizalayarak yapısını ve işlevini tahmin edebilirler.
Homoloji aramaları, patojen proteinlerini insan proteinleriyle karşılaştırarak umut verici ilaç hedeflerini ortaya çıkarabilir veya bir hastanın proteinini sağlıklı bir versiyonuyla karşılaştırarak hastalığa yol açan mutasyonları tespit edebilir. Ancak, genomik ve metagenomik verilerin hızlıca artışı, geleneksel hizalama araçlarının sınırlarını zorlamaktadır.
Bu yazıda, son zamanlarda yaşanan gelişmelerin protein bilimini nasıl hızlandırdığına ve GPU destekli hizalamanın AI destekli ilaç keşfi, yapısal tahmin ve protein tasarımında sağladığı olağanüstü hıza odaklanılacaktır.
Protein Dizi Hizalaması Bilimsel İçgörüye Katkı Sağlıyor
Dizi hizalaması teknik olarak karmaşık görünebilir, ancak önemi oldukça açıktır: Bilim insanları protein dizilerini karşılaştırarak benzerlikler bulurlar. Benzer diziler, çoğu zaman benzer işlev veya yapısal özellikler taşıdığını gösterir. Homoloji çıkarımı bu ilkeye dayanıyor: Eğer Protein A, Protein B’ye benziyorsa, muhtemelen benzer biyolojik rolleri vardır.
Protein dizi hizalaması, korunmuş alanları belirleme, protein işlevlerini tahmin etme ve hastalığa yol açabilecek şüpheli mutasyonları tespit etme gibi birçok alanda kritik öneme sahiptir. Dizi hizalamalarındaki evrimsel bilgiler, ilaç hedeflerinin seçimi ve optimize edilmesinde de yol gösterici olmaktadır.
İnsan proteomunun büyüklüğü son derece karmaşıktır. Tipik bir memeli hücresi yaklaşık 10 milyar protein molekülü içerir ve bu proteinler hücre başına 106 çeşitlilik gösterir. Vücut sıvıları, en bol ve en az bulunan proteinler arasında 1010 katlık bir fark ortaya koyar. Bu karmaşıklık, kapsamlı proteomik analizlerin gerçekleştirilmesini zorlaştırmaktadır.
Protein etkileşimlerini haritalamak ise daha da zordur. İnsan proteomunda neredeyse 200 milyon olası çift yönlü etkileşim bulunmasına rağmen, yalnızca yaklaşık 53,000’i deneysel olarak onaylanmıştır, bu durum haritalamayı samanlıkta iğne aramak gibi zor bir hale getirir.
De novo protein tasarımı, bu zorluklardan en karmaşık olanıdır ve astronomik bir arama uzayını (N uzunluğundaki her bir protein için 20N dizisi) kapsar. Bu, katlama ve işlev optimizasyonu ile ilgili bir NP-zor bir iştir. AI ve otomasyon alanındaki ilerlemeler başarı oranlarını önemli ölçüde artırmıştır—%10’dan %30-50’ye kadar çeşitli tasarım sınıfları için—ancak deneysel doğrulama kaynak açısından yoğun olmayı sürdürmekte, çoğu zaman döngüsel test ve iyileştirme gerektirmektedir. Son gelişmeler ilerlemeyi hızlandırsa da bu temel sorunlar, yapısal biyolojinin ön saflarında kalmaktadır.
BLAST’tan MMseqs2’ye Hizalamaların Evrimi
Biyoinformatikçiler, dizi hizalamasını hızlandırmak için giderek daha verimli algoritmalar geliştirmiştir. 1990’larda BLAST, arama hızlarında devrim yarattı, ancak veri büyüklüğündeki artışlarla mücadele edemeyince 2010’larda DIAMOND ve MMseqs2 geliştirildi.
MMseqs2, PSI-BLAST’tan daha iyi duyarlılık sağlar ve 400 kat daha hızlı çalışır. Üç iterasyonla yapılan profil aramalarda, MMseqs2, PSI-BLAST’tan 433 kat daha hızlıdır ve aynı zamanda oldukça yüksek bir duyarlılık sergilemektedir.
MMseqs2 ve DIAMOND, artık genomi anotasyonu ve ilaç keşfi alanında yaygın bir şekilde kullanılmakta ve bir zamanlar haftalar süren hesaplamaları birkaç saat içinde sonuçlandırmaktadır. Ancak verilerin hacmi arttıkça, en hızlı CPU tabanlı araçlar bile sınırlara ulaşmakta, bu da GPU hızlandırmasına yönelmeyi zorunlu kılmaktadır. AI’deki ilerlemelere rağmen, protein hizalamaları hâlâ kritik öneme sahiptir. AlphaFold2 gibi derin öğrenme modelleri, protein yapılarını tahmin etmek için çoklu dizi hizalamalarına (MSA’lar) ihtiyaç duyar, bu da hızlı ve ölçeklenebilir dizi arama yöntemlerinin önemini göstermektedir (Tablo 1).
| MMseqs2 Kullanım Durumu | Örnekler | Referanslar |
| Protein yapısı tahmini için gelişmiş ve kademeli MSA aramaları | 1. ColabFold 2. RoseTTAFold ve ColabFold Arama 3. OpenFold ve ColabFold Arama |
AlphaFold ile Yüksek Doğru Protein Yapısı Tahmini
ColabFold: Protein Katlamayı Herkese Erişilebilir Hale Getiriyor Protein Yapıları ve Etkileşimlerinin Doğru Tahmini Üç İz Track Sinir Ağı Kullanılarak Yapılıyor |
| Tekrarlayan veya homolog dizileri filtreleme | ADOPT: İçsel olarak düzensiz protein alanlarını tanımlar | ADOPT: Derin Bidirektör Transformerlar Yoluyla İçsel Protein Düzensizliği Tahmini |
| Protein etkileşim analizi için kümeleme | SENSE-PPI | SENSE-PPI, Genom Ölçeğinde İçinde, Arasında ve Türler Arasında Etkileşim Ağlarını Yeniden İnşa Ediyor |
Daha hızlı MSA’nın derin öğrenmeye dayalı iş akışlarında sağladığı potansiyel fayda büyüktür. MSA arama, genellikle hesaplama açısından maliyetli olup, tahmin ve eğitim sürelerini önemli ölçüde aşabilmektedir. Örneğin, AlphaFold2 ve OpenFold için tahmin süreleri, MSA araması tarafından domine edilmektedir (toplam zamanın ~%70-90’ı bu alana aittir), bu da AlphaFold ve OpenFold için %57 ve %51’lik maliyet tasarrufu sağlamaktadır.
MMseqs2-GPU ve Hızda Bir Sonraki Atılım
MMseqs2-GPU, çoklu dizi hizalamalarını CUDA üzerinde gerçekleştiren novel GPU spesifik hızlandırmalar sunmaktadır. MMseqs2-GPU’yu geliştiren araştırma ekibi, Seul Ulusal Üniversitesi, Johannes Gutenberg Üniversitesi Mainz ve NVIDIA’dan oluşmaktadır. Ekip, CPU tabanlı ön filtre yerine kısmen boşluk bırakmayan bir algoritma oluşturmuştur.
Basitçe ifade etmek gerekirse, BLAST ve MMseqs2’nin CPU ile gerçekleştirdiği gibi kısa eşleşen alt dizeleri (k-mer) aramak yerine, GPU versiyonu, hizalamaları boşluk bırakmadan doğrudan puanlayan son derece paralel bir algoritma kullanır. Bu yöntem, bellek darboğazlarını önlemek ve aynı anda binlerce GPU çekirdeğini meşgul tutmak için tasarlanmıştır. Ön filtre, umut verici eşleşmeleri bulduktan sonra, GPU ayrıca bu bulgular için daha hassas boşluk bırakmalı hizalamalar gerçekleştirebilmektedir (optimize edilmiş Smith-Waterman algoritması kullanılarak).
Bu gelişmeyi anlamak için MMseqs2 GPU teknik raporu‘ndan bazı temel performans karşılaştırmalarını inceleyebiliriz:
- MMseqs2-GPU, boşluk bırakmayan filtreleme işlemlerinde tek bir GPU üzerinde 100 TCUPS (trilyon hücre güncellemeleri saniyede) başarıyor ve önceki hızlandırma yöntemlerini 1-2 kat daha aşar.
- Tek bir NVIDIA L40S GPU’da, MMseqs2-GPU, 128 çekirdekli bir CPU’da çalışan MMseqs2 k-mer’den 20 kat daha hızlı ve 71 kat daha ucuzdur.
- ColabFold’de, MMseqs2-GPU, AlphaFold2’ye karşı JackHMMER kullandığında yapı tahminini 23 kat hızlandırmaktadır, aynı zamanda eşdeğer tahmin doğruluğunu korumaktadır.
- Protein yapı hizalaması için Foldseek’te, MMseqs2-GPU CPU versiyonuna göre 27 kat hız artışı sağlamaktadır.
- MMseqs2-GPU, uygun maliyetli GPU’larda bile, örneğin NVIDIA L4 gibi, JackHMMER’a göre UniRef90’da arama yaparken on kat hızlıdır.
Dizi Hizalaması ve Protein Yapısı Tahmini
MMseqs2-GPU’nın ilaç keşif iş akışındaki gerçek bir sorunu nasıl çözdüğüne dair bir örnek altında, dizisel hizalamanın AI destekli hedef yapı tahmini üzerindeki rolü açıklanacaktır. Burada, MMSeqs2-GPU (MSA-Arama) ve OpenFold modeli, birbirleriyle etkileşim içinde çalışan konteynerleştirilmiş NVIDIA NIM mikro hizmetleri olarak paketlenmiştir (Şekil 2).
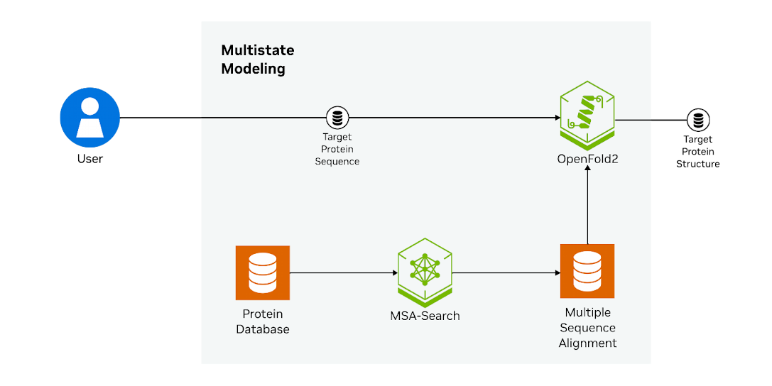
Örneğin, MSA-Arama NIM ve OpenFold2 NIM birlikte kullanılarak ilgilenilen bir proteinin çoklu konformasyonel durumları modellenebilir. İş akışı aşağıdaki adımları içerir:
- Sorgu → Hizalama: FASTA dizisini MSA-Arama NIM‘a gönder → Bir A3M MSA ve bir şablon-hiti HHR dosyası al.
- Şablonları seç: İki PDB ID’si seç (açık vs kapalı) ve bireysel HHR bloklarını çıkart.
- Her durumu tahmin et: OpenFold 2 NIM‘i iki kez çağır, aynı A3M’yi ama her seferinde farklı bir HHR dilimi geçir.
- Sonuçları kaydet: İki dönen PDB dizesini hClpP_active.pdb ve hClpP_inactive.pdb olarak kaydet.
Aşağıdaki kod parçası, bu adımların nasıl gerçekleştirileceğini gösterir; burada insan hCLpP proteini bir örnek olarak kullanılmıştır:
"""
Örnek: İnsan ClpP (hClpP) için iki konformasyonu modelleme
"""
import os, json, requests, pathlib
# ------------------------------------------------------------------
# Yapılandırma
# ------------------------------------------------------------------
API_KEY = os.environ["NIM_API_KEY"]
HEADERS = {"Authorization": f"Bear!er {API_KEY}", "Content-Type": "application/json"}
MSA_URL = "http://localhost:8000/biology/colabfold/msa-search/predict"
OF2_URL = "http://localhost:8000/biology/openfold/openfold2/predict-structure-from-msa-and-template"
SEQ = """>hClpP_HUMAN
MARGKIIGELASKKKVEAMAAKLAEAG... (FASTA açıklık için kısaltılmıştır)"""
# ------------------------------------------------------------------
# 1) MSA-Arama NIM ile GPU-MMseqs2 çalıştır
# ------------------------------------------------------------------
msa_payload = {
"sequence": SEQ,
"databases": ["uniref90-2024_02", "pdb70"],
"return_templates": True # hizmete, HHR formatında bir hit listesi oluşturmasını söyler
}
msa_resp = requests.post(MSA_URL, headers=HEADERS,
data=json.dumps(msa_payload), timeout=900)
msa_resp.raise_for_status()
msa = msa_resp.json()
a3m_alignment = msa["alignments"]["uniref90-2024_02"]["a3m"]["alignment"]
# Yardımcı: İki PDB şablonu seç
ACTIVE_PDB = "7DKF" # hClpP aktif/açık
INACTIVE_PDB = "7D7G" # hClpP inaktif/kapatılmış
hhr_all = msa["templates"]["pdb70"]["hhr"]["alignment"] # tam HHR metni
def slice_hhr_for(pdb_id: str, hhr_text: str) -> str:
"""Tek bir şablon PDB vuruşu için bir HHR minimal bloğunu döndür."""
keep = []
write = False
for line in hhr_text.splitlines():
if line.startswith(">PDBID:") and pdb_id in line:
write = True
elif line.startswith(">PDBID:") and write:
break
if write: keep.append(line)
return "n".join(keep)
hhr_active = slice_hhr_for(ACTIVE_PDB, hhr_all)
hhr_inactive = slice_hhr_for(INACTIVE_PDB, hhr_all)
# ------------------------------------------------------------------
# 2) Aktivite konformasyonunu OpenFold 2 ile tahmin et
# ------------------------------------------------------------------
def run_openfold2(hhr_block: str, tag: str) -> pathlib.Path:
payload = {
"sequence": SEQ,
"alignments": {
"uniref90-2024_02": {
"a3m": {"alignment": a3m_alignment, "format": "a3m"}
}
},
"templates": {
"pdb70": {
"hhr": {"alignment": hhr_block, "format": "hhr"}
}
},
"selected_models": [1] # hız için tek model çalıştır; topluluk için hariç tut
}
r = requests.post(OF2_URL, headers=HEADERS,
data=json.dumps(payload), timeout=1800)
r.raise_for_status()
pdb_text = r.json()["predictions"][0]["structure"]
out = pathlib.Path(f"hClpP_{tag}.pdb")
out.write_text(pdb_text)
print(f"{out} yazıldı")
return out
active_pdb = run_openfold2(hhr_active, "active")
inactive_pdb = run_openfold2(hhr_inactive, "inactive")
Gelecek Yönelimleri: GPU Destekli Biyoinformatik
Dizi hizalamanındaki ilerlemeler, BLAST’tan MMseqs2-GPU’ya kadar, protein biliminde devrim yaratarak işlev, evrim ve ilaç keşfine yönelik daha hızlı içgörüler sağlamaktadır.
Bu araç, AI model geliştirme döngüsünün önemli bir parçası olan sentetik veri setleri üretiminde ve test zamanı ölçeklendirme ile gerçek zamanlı tahminler için hızlandırılmış çıkarımda kullanılmaktadır. MMseqs2-GPU, Basecamp Research, VantAI ve Iambic Therapeutics gibi önde gelen şirketler tarafından yaygın bir şekilde benimsenmiştir.
AI destekli modellerin tahmin iş akışlarına hizalamayı entegre etmesiyle, GPU hızlandırması moleküler araştırmaları yeniden tanımlamaktadır. AI, HPC ve biyoinformatik entegrasyonunun büyük kırılmalara imza atması ve tıp ile biyoteknolojideki keşifleri hızlandırması beklenmektedir. NVIDIA BioNeMo Blueprint ile biyolojik bağlayıcı tasarımı hakkında daha fazla bilgi edinin.
MMSeqs2-GPU’yu NVIDIA MSA-Arama NIM olarak deneyebilirsiniz; ayrıca OpenFold’u NVIDIA OpenFold2 NIM olarak test edebilirsiniz.