Topluluk tespiti algoritmaları, verileri anlamada önemli bir rol oynayarak ağlardaki ilişkili varlıkların gizli gruplarını belirlemeye yardımcı olur. Sosyal ağ analizi, öneri sistemleri, GraphRAG, genomik ve daha birçok alan, topluluk tespitine dayanır. Ancak, Python’da çalışan veri bilimcileri için, grafik verilerini etkin bir şekilde analiz etme yeteneği, büyüyen veri setleri için yanıt veren, ölçeklenebilir topluluk tespit sistemleri oluşturma açısından bir sorun teşkil edebilir.
Günümüzde birçok topluluk tespit algoritması bulunmakla birlikte, Leiden algoritması, veri bilimcileri için öne çıkan bir çözüm haline gelmiştir. Ayrıca, büyük ölçekli grafiklerde, bu bir zamanlar pahalı olan işlem, cuGraph ve onun GPU hızlandırmalı Leiden uygulaması sayesinde artık çok daha hızlıdır. cuGraph’ın Leiden uygulaması, karşılaştırılabilir CPU alternatiflerine göre %47’ye kadar daha hızlı sonuçlar sunar. Bu performans, Python iş akışlarınızda cuGraph Python kütüphanesi veya NetworkX kütüphanesi aracılığıyla nx-cugraph backend’i kullanılarak kolayca erişilebilir.
Bu yazı, Leiden algoritmasının nerelerde kullanılabileceğini ve gerçek dünya veri boyutları için cuGraph ile nasıl hızlandırılacağını göstermektedir. Ayrıca, cuGraph Leiden performansının diğer Python kütüphanelerine karşı kıyasını ve daha büyük ölçekli genomik verilerde GPU hızlandırmalı Leiden örneğini paylaşacağız.
Leiden Nedir?
Leiden, popüler Louvain algoritmasının bir modifikasyonu olarak geliştirilmiştir. Louvain gibi, bir ağı topluluklara ayırmayı ve bir kalite fonksiyonu olan modülerliği optimize etmeyi amaçlar. Ancak, Leiden’in Louvain’den önemli bir farkı vardır: Louvain’in döndürdüğü topluluklar, bazen bağlantısız olabilen zayıf şekilde bağlı topluluklar olabilir. Leiden, bu sorunu aşmak için bir ara iyileştirme aşaması ekleyerek sonuçlanan toplulukların iyi bağlantılı olmasını garanti eder; bu da onu geniş bir uygulama yelpazesinin popüler bir tercihi haline getirmektedir. Leiden, hızla Louvain’e alternatif standart haline gelmektedir.
Leiden Nerelerde Kullanılmaktadır?
Aşağıda, Leiden gibi topluluk tespit tekniklerini kullanan ve sürekli büyüyen gerçek dünya veri boyutlarından etkilenen birkaç alan örneği yer almaktadır:
- Sosyal ağ analizi: Toplulukları tanımlamak, benzer ilgi alanlarına sahip kullanıcı gruplarını ortaya çıkararak hedefli reklamcılık, öneriler ve bilgi yayılımı çalışmalarını kolaylaştırabilir.
- Öneri sistemleri: Kullanıcıları veya öğeleri etkileşimlerine dayalı olarak topluluklara ayırmak, öneri sistemlerinin daha doğru ve kişiselleştirilmiş öneriler sunmasına olanak tanır.
- Sahtekarlık tespiti: Mali ağlarda sahte hesapların veya şüpheli işlemlerin topluluklarını belirleyerek kurumların hızlı bir şekilde sahtekarlığı tespit etmesi ve incelemesi sağlanır.
- Graf temelli geri alma artı üretim (GraphRAG): GraphRAG, bir bilgi grafiğinden ilgili bilgileri alarak bir LLM’ye (Büyük Dil Modeli) daha iyi bir bağlam sağlamak için kullanılır. Leiden, bilgi grafiğindeki en uygun düğüm eşleştirmelerini kolaylaştırmak için bilgi kategorileri oluşturmak amacıyla sıkça kullanılır.
- Genomik: Leiden, tek hücre genomik verilerini analiz ederken benzer gen ekspresyon profillerine sahip hücre gruplarını tanımlamak için kullanılır.
cuGraph ile GPU Güçlendirilmiş Leiden Nasıl Karşılaştırılır?
Pythonda geliştiricilere sunulan çeşitli Leiden uygulamaları, 3.8 milyon düğüm ve 16.5 milyon kenar içeren bir patent alıntı grafiği kullanılarak karşılaştırılmıştır; bu grafikte Leiden tarafından tanımlanan topluluklar, ilişkili teknolojileri temsil etmektedir. Şekil 1, saniye cinsinden çalışma süresi ile birlikte belirlenen eşsiz topluluk sayısını göstermektedir.
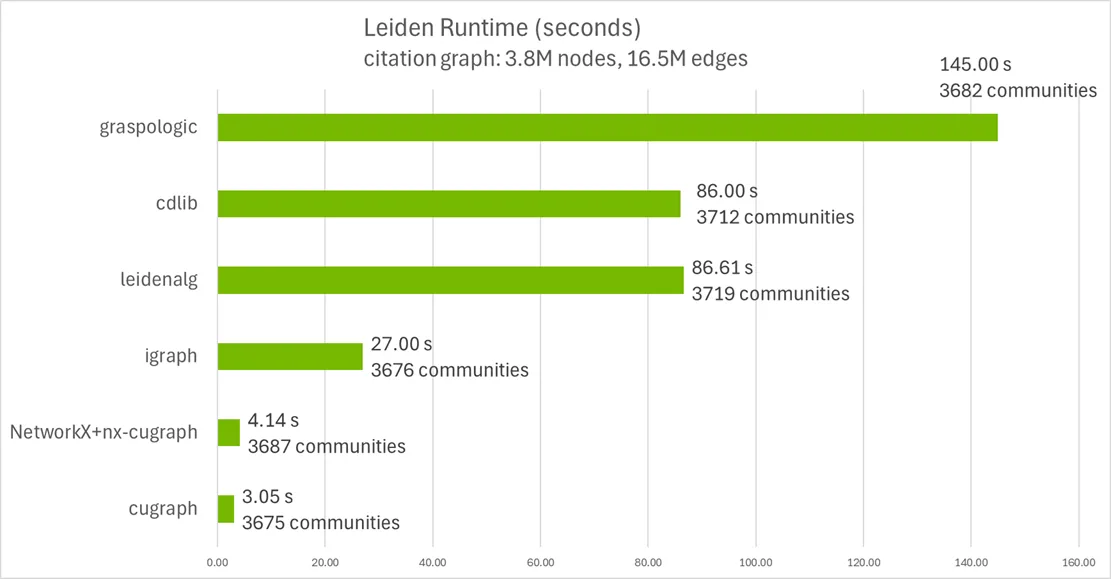
Yazılım: NetworkX 3.5, cugraph/nx-cugraph 25.10; CPU: Intel Xeon Platinum 8480CL 2TB RAM; GPU: NVIDIA H100 80GB RAM
Leiden uygulamaları bir rastgele sayı üreteci kullandığı için, döndürülen topluluklar deterministik değildir ve çalıştırmalar arasında biraz farklılık gösterebilir. Topluluk sayısının gösterimi, tüm sonuçların yaklaşık olarak eşit olduğunu belirtmek içindir. Birçok uygulama, daha büyük veya daha küçük topluluk boyutları için ayarlamalar yapmaya olanak tanıyan parametreler sunar. Her uygulama, mümkün olduğunda varsayılan parametre değerleri ile çağrılmıştır. Bu kıyasların kaynak kodu, rapidsai/cugraph GitHub deposunda bulunabilir.
Şekil 1’de gösterildiği gibi, cuGraph GPU hızlandırmalı Leiden uygulaması, aynı alıntı grafiği üzerinde igraph‘ın uygulamasına göre 8.8 kat ve graspologic‘in uygulamasına göre 47.5 kat daha hızlıdır. Yüksek performansın yanı sıra, cuGraph ayrıca mevcut Python veri bilimi iş akışlarıyla uyumluluk, kullanım kolaylığı ve esneklik de sunar. Projeniz için en uygun olanı seçmenize yardımcı olmak adına Tablo 1, her kütüphanenin temel özelliklerini sunmaktadır. Leiden ve birçok diğer grafik algoritması her birinde mevcuttur.
| Hız | Kullanım Kolaylığı | Bağımlılıklar | NetworkX avantajları: CPU geri dönme, esnek grafik nesnesi, popüler API, yüzlerce algoritma, grafik görselleştirme vb. | Çoklu GPU desteği | cuDF ve Dask desteği |
| NetworkX ve nx-cugraph | Hızlı | En Kolay | Az | ✔ | |
| cuGraph | Daha Hızlı | Kolay | Daha Fazla, cuDF ve Dask dahil | ✔ | ✔ |
Ayrıntılı kurulum talimatları için RAPIDS Kurulum kılavuzu‘na bakın. Hızla başlamak için pip veya Conda kullanarak RAPIDS sürüm seçiciyi kullanın.
Genomik Verilerle NetworkX ve nx-cugraph Nasıl Kullanılır?
Genomik veri setleri oldukça büyüktür ve DNA dizileme maliyetlerindeki son derece dramatik düşüş nedeniyle patlama gibi bir artış göstermektedir. NetworkX, birçok alandaki veri bilimcileri arasında büyük bir üne sahiptir; ancak tamamen Python’da yazılmış olması, çoğu genomik veri setinin onun için çok büyük olmasına neden olmaktadır. Bu nedenle, bilim insanları başka bir kütüphane öğrenmek ve entegrasyon yapmak zorunda kalmaktadır. Neyse ki, NetworkX, nx-cugraph backend’ini etkinleştirerek GPU hızlandırılabilir. Bu da veri bilimcilerinin büyük veri setleriyle çalışmaya devam etmelerini sağlamaktadır.
Büyük ölçekli genomik veriler üzerinde GPU hızlandırmanın faydasını göstermek için, güzel bir örnek hazırlandı. Gene ekspresyon verilerini okumak, genleri ekspresyon korelasyon değerlerine göre bağlayan kenarlarla grafik oluşturmak, Leiden’i çalıştırarak fonksiyonel olarak ilişkili gen gruplarını tanımlamak ve toplulukları görsel olarak incelemek için grafik çizimi yapılmaktadır. Tam kaynak kodu, rapidsai/nx-cugraph GitHub deposunda bulunabilir. Bu örnek, genellikle genomik çalışmalarda kullanılan topluluk tespiti için Leiden veya Louvain’ın kullanıldığı bir işlemi temsil etmektedir, ancak tipik bir genomik iş akışını temsil etmek üzere tasarlanmamıştır.
Gen ekspresyon analizi verisi kullanılarak oluşturulan grafik, 14.7K düğüm ve 83.8 milyon kenar içermektedir. Aşağıdaki kod, nx-cugraph kullanarak Leiden’i çalıştıracak, ancak nx-cugraph mevcut olmadığında NetworkX’un Louvain uygulamasına geri dönecektir.
Leiden, şu anda nx-cugraph sayesinde mevcut olan ancak NetworkX tarafından alternatif bir uygulaması olmayan tek algoritmadır. Bu yüzden, bu iş akışı, bir GPU’nun bulunmadığı durumlarda NetworkX kullanmaya devam etmek isteyen kullanıcılar için makul bir karşılaştırma sunan Louvain’i kullanmaktadır.
nx-cugraph etkinleştirildiğinde, NetworkX dört topluluğu 4 saniyeden daha kısa bir sürede tanımladı. Ancak, NetworkX’un Louvain uygulamasına geri dönüldüğünde, sonuçların neredeyse eşit olduğunu (her iki algoritmanın da deterministik olmaması nedeniyle tolerans dahilinde) görmekle birlikte, performansın dramatik bir şekilde yavaşladığı ve neredeyse 21 dakika sürdüğü görülmektedir. Ayrıca, Louvain kullanıldığı için, sonuçlanan toplulukların iyi bağlantılı olacağı garantisi yoktur.
Bu durum, NetworkX ve nx-cugraph‘ın daha yüksek kaliteli sonuçları 315 kat daha hızlı sunmasını sağlamaktadır.
Leiden veya Louvain’in varlığına dayanarak çalıştırmak için aşağıdaki kodu kullanın:
%%time
try:
communities = nx.community.leiden_communities(G)
except NotImplementedError:
print("Leiden mevcut değil (cuGraph backend'i etkinleştirildi mi?), Louvain kullanılıyor.")
communities = nx.community.louvain_communities(G)
num_communities = len(communities)
print(f"Topluluk sayısı: {num_communities}")

Yazılım: NetworkX 3.5, cugraph/nx-cugraph 25.10; CPU: Intel Xeon Gold 6128 CPU @ 3.40 GHz 48 GB RAM; GPU: NVIDIA Quadro RTX 8000 48 GB RAM
Ayrıca, topluluk bazında graf düğümlerini renklendirmek ve grafik çizmek, NetworkX’te oldukça kolaydır (Şekil 3).
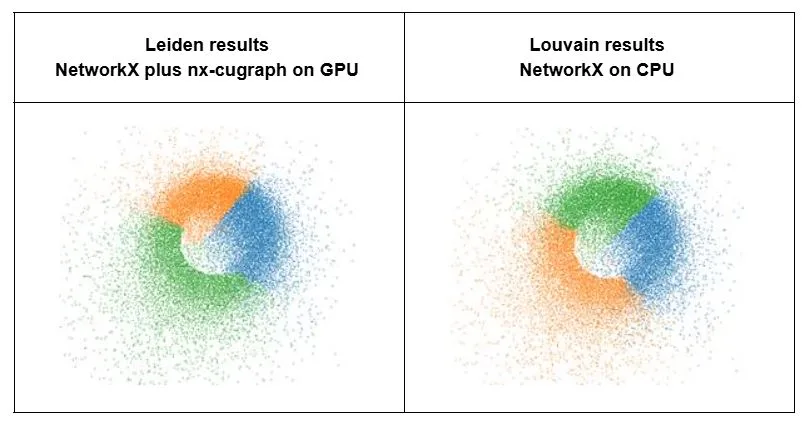
NetworkX, Leiden için CPU desteğini eklediğinde, ya yerel Python uygulaması olarak ya da ayrı bir CPU backend’i olarak, kullanıcılar tek bir “taşınabilir” işlev çağrısıyla, GPU olmayan platformlarda çalışabilmesi için herhangi bir kod değişikliğine ihtiyaç duymadan yararlanabilecektir.
GPU Hızlandırmalı Leiden Kütüphanesi İçin Özel Araçlar
Önceki örnek, nx-cugraph’ın gerçek dünya genomik verilerde yaygın olarak kullanılan algoritmaları GPU hızlandırması ile nasıl hızlandırabileceğini basit şekilde göstermek amacıyla tasarlanmıştır. Daha gerçekçi ve özel örnekleri keşfetmek için, RAPIDS-singlecell projesine göz atın. Bu proje, genomik problemler için özel olarak tasarlanmış bir kütüphanedir.
RAPIDS-singlecell, popüler Scanpy kütüphanesine dayanan bir scverse temel paketidir. AnnData uyumlu bir API’yi destekler ve büyük veri setlerinde tek hücre analizi için optimize edilmiştir. RAPIDS-singlecell’ın ölçeklenebilirliğindeki etkileyici hız cuGraph ve diğer CUDA-X DS kütüphanelerinin Leiden ve birçok diğer algoritma için GPU hızlandırması sağlamasıyla elde edilmektedir. Daha fazla bilgi için, RAPIDS-singlecell ile Milyar Hücre Analizine ve Biyolojik Atılımlara Doğru başlıklı çalışmayı inceleyebilirsiniz.
GPU Güçlendirilmiş Leiden Çalışmaları Çalıştırmaya Başlayın
cuGraph, GPU hızlandırmalı Leiden uygulaması ile topluluk tespitinde sınıfının en iyisi performansı sunar. Bu, veri bilimcilerin Python’da cuGraph Python kütüphanesi veya popüler ve esnek NetworkX kütüphanesi üzerinden nx-cugraph backend’i aracılığıyla erişebileceği bir atanmadır. %47’ye kadar daha hızlı performans, belki daha fazlası, genomik gibi birçok uygulamanın topluluk tespitine ihtiyaç duyan verilerini ölçeklendirmesine ve daha büyük sorunları çok daha kısa sürede çözmesine olanak tanımaktadır.
Başlamak için, RAPIDS Kurulum kılavuzu‘na göz atın ya da rapidsai/cugraph veya rapidsai/nx-cugraph GitHub depolarına göz atarak GPU güçlendirilmiş Leiden iş akışlarınızı çalıştırın.