Uzun yıllar boyunca, biyolojinin en derin sırlarından biri, amino asitlerin nasıl kıvrılarak yaşamın karmaşık mimarisine dönüştüğünü anlamaktı. Araştırmacılar, bir sonuca ulaşmak için titiz simülasyonlar ve istatistiksel modeller geliştirdiler, ancak bu alanda kapsamlı bir tahmin yapmayı başaramadılar.
Sonunda, derin öğrenme her şeyi değiştirdi. AI, evrimin dilini doğrudan dizilim verilerinden öğrenerek, moleküler formun gizli kurallarını ortaya çıkardı ve yapısal tahmini bir sanat dalından mühendislik disiplini haline dönüştürdü.
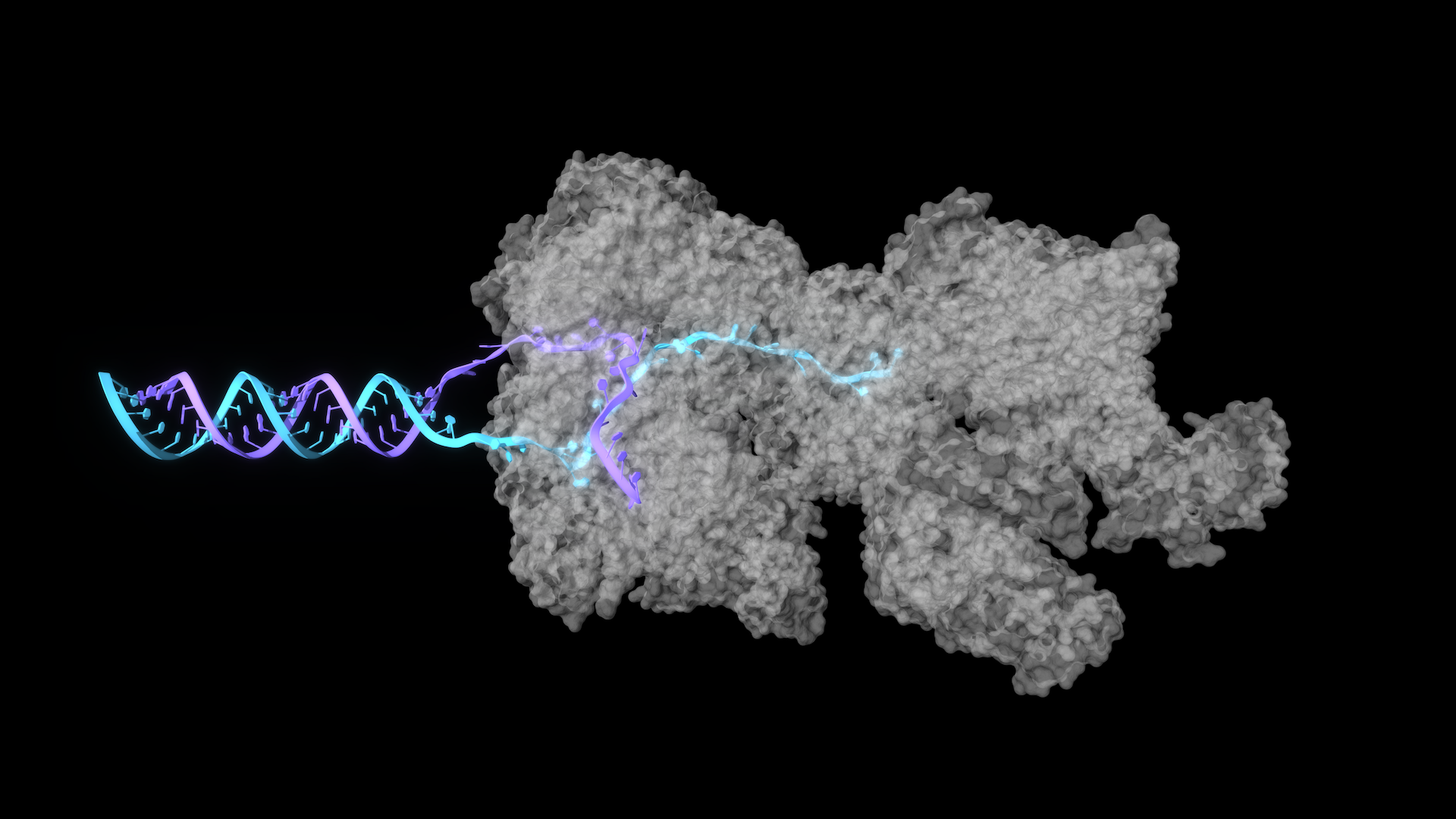
Bugün bu dönüşüm yeni bir aşamaya ulaşıyor. OpenFold3, üretime hazır protein AI’sını NVIDIA ekosistemine getiriyor ve açık bilimi kurumsal düzeyde performansla birleştiriyor. OpenFold Konsorsiyumu tarafından geliştirilen ve NVIDIA tarafından desteklenen OpenFold3, yapı tahminini tekli proteinlerden çok zincirli komplekslere, nükleik asitlere ve küçük molekül ligandlarına kadar genişletiyor; böylece biyolojik etkileşimin tüm dilbilgisini modelliyor.
Öne Çıkan Özellikler
NVIDIA cuEquivariance ile simetriye duyarlı GPU hızlandırması, MMseqs2-GPU ile hızlı dizilim araması ve NVIDIA FLARE ile federatif eğitim sayesinde OpenFold3, dünya genelindeki biyofarma ve biyoteknoloji ekipleri için benzersiz hız, ölçek ve gizlilik koruma sunuyor.
OpenFold3 artık mevcut ve NVIDIA NIM ile ek hızlandırma sağlıyor. Bu yazı, OpenFold3 NIM’i yapı tahmini çalışmalarınız için nasıl kullanacağınızı açıklıyor.
Başlamak İçin Hızlı Bağlantılar
Gereksinimler
- Docker destekleyen bir işletim sistemi ile başlayın.
- Docker‘ı kurun (minimum sürüm: 23.0.1).
- NVIDIA sürücüleri‘ni yükleyin (minimum sürüm: 580 – CUDA: 13.0).
- NVIDIA Container Toolkit‘ı kurun (minimum sürüm: 1.13.5).
- OpenFold3 NIM belgelerini okuyun.
OpenFold3 NIM ile Yapı Tahmini
OpenFold3 NIM sayesinde yapı tahmini, yalnızca birkaç adımda prototip aşamasından üretime geçebilir.
1. Adım: Modeli Erişin
OpenFold3 NIM, build.nvidia.com üzerinden erişilebilir. Konteyneri yerel olarak, bir kümede veya yönetilen bir NIM hizmeti olarak dağıtabilirsiniz.
Docker komutunu kullanarak açma:
docker pull nvcr.io/nim/openfold/openfold3:latest
export LOCAL_NIM_CACHE=~/.cache/nim
export NGC_API_KEY=
docker run --rm --name openfold3
--runtime=nvidia
--gpus 'device=0'
-p 8000:8000
-e NGC_API_KEY
-v $LOCAL_NIM_CACHE:/opt/nim/.cache
--shm-size=16g
nvcr.io/nim/openfold/openfold3:latest
2. Adım: Yapı Tahmin İşlemine Gönderin
Dağıtım yapıldıktan sonra API ile standart REST çağrıları veya Python istemcileri kullanarak etkileşimde bulunabilirsiniz:
#!/usr/bin/env python3
import requests
import os
import json
from pathlib import Path
output_file = "output.json"
url = "http://localhost:8000/biology/openfold/openfold3/predict"
protein_sequence = "MGREEPLNHVEAERQRREKLNQRFYALRAVVPNVSKMDKASLLGDAIAYINELKSKVVKTESEKLQIKNQLEEVKLELAGRLEHHHHHH"
msa_alignment_csv = "key,sequencen-1,MGREEPLNHVEAERQRREKLNQRFYALRAVVPNVSKMDKASLLGDAIAYINELKSKVVKTESEKLQIKNQLEEVKLELAGRLEHHHHHH"
dna_sequence_b = "AGGAACACGTGACCC"
dna_sequence_c = "TGGGTCACGTGTTCC"
data = {
"request_id": "5GNJ",
"inputs": [
{
"input_id": "5GNJ",
"molecules": [
{
"type": "protein",
"id": "A",
"sequence": protein_sequence,
"msa": {
"main_db": {
"csv": {
"alignment": msa_alignment_csv,
"format": "csv",
}
}
}
},
{
"type": "dna",
"id": "B",
"sequence": dna_sequence_b
},
{
"type": "dna",
"id": "C",
"sequence": dna_sequence_c
}
],
"output_format": "pdb"
}
]
}
r = requests.post(url=url, json=data)
print(r, "output.json olarak kaydediliyor:n", r.text[:200], "...")
Path(output_file).write_text(r.text)
Tahminler, 3D koordinatlar (PDB/mmCIF) ve pLDDT, pTM gibi güvenilirlik ölçümleri ile birlikte sunulmakta ve NVIDIA H100 Tensor Core GPU’lar üzerinde saniyeler içinde sağlanmaktadır.
Yeni Bir Açık Standart: Protein Yapı Tahmini
OpenFold Konsorsiyumu, Bayer, Bristol Myers Squibb, Johnson & Johnson, Novo Nordisk, Outpace Bio ve diğerlerini içeren sanayi odaklı bir koalisyon, açık, tekrarlanabilir modelleme sistemlerini ilerletmede önemli bir rol oynamıştır.
OpenFold3, konsorsiyumun en büyük başarısıdır. Bu model, yapı tahminini multimere, protein–DNA/RNA komplekslerine ve ligand bağlı bileşimlere genişleterek, en iyi açık kaynak modellerin doğruluğunu yakalamaktadır.
Özellikle, OpenFold3, protein–nükleik asit benchmark’larında AlphaFold3 performansına ulaşarak, daha önceki modellerin geride kaldığı bir alanda önemli bir başarı sağlamaktadır. Linux Vakfı açık model tanımına göre Sınıf 1 açık kaynak sistemi olarak sınıflandırılarak, tam şeffaflık ve tekrarlanabilirlik sağlamaktadır.
Açık Bilim ve Kurumsal Güvenilirlik
OpenFold3, NVIDIA hızlandırılmış AI hesaplama yığını için optimize edilmiştir ve aşağıdaki bileşenleri içerir:
- cuEquivariance: 3D simetri işlemleri için fizik tabanlı hızlandırma.
- MMseqs2-GPU: Bu GPU yerel çoklu dizilim aracı ile uyumlu.
- NVIDIA FLARE: Veri paylaşmadan kurumsal modeller üzerinde işbirliği için uygun.
Bu entegrasyonlar, OpenFold3 NIM’i hem geliştiriciye erişilebilir, hem de kurumsal düzeyde dağıtılabilir hale getirir; bu, yerel, hibrit ve bulut ortamları için bir servis imkânı sunar. NVIDIA TensorRT büyük multimeler ve nükleik asit kompleksleri için %80’e kadar daha hızlı çıkarım sağlar.
OpenFold3, Apheris ve SandboxAQ tarafından güvenli federatif iş akışlarında doğrulanmış olup, küresel farmasötik Ar-Ge ortamlarında ölçeklenme kapasitesini göstermektedir. Federatif süreçlerle ortaklar, hassas verilerin paylaşımına gerek kalmadan, özel veriler üzerinde fine-tune süreçleri gerçekleştirebilir.
OpenFold3, Linux Vakfı açık model tanımına göre Sınıf 1 açık bir sistem olarak sınıflandırıldığından, hızlı büyüyen bir katkıda bulunma ve benchmark topluluğundan faydalanmaktadır. Bu da sürekli iyileşme ve uzun vadeli güvenilirlik sağlar.
Açık Protein AI Geleceği İnşası
OpenFold3, yalnızca bir model değildir; aynı zamanda protein AI’sının gelecek on yılı için bir temeldir. OpenFold Konsoriyumu‘ndaki 40’tan fazla kurumun, açık kaynak biliminin, hızlandırılmış hesaplamanın ve federatif işbirliğin birleşimini yansıtarak, dünya genelindeki araştırmacılar tarafından kullanılan araçların aynı zamanda kurumsal güvenilirlik ve güvenlik standartlarını karşılamasını sağlamaktadır.
Özel teşekkürlerimi OpenFold Konsorsiyumu’na ve SandboxAQ, Apheris gibi ortaklara, moleküler bilimler için açık, hızlandırılmış AI’yi ilerletmedeki işbirlikleri için sunuyorum.