Biyolojik Modeller İçin Transformer Motorunun Hızlandırılması
Trilyonlarca parametreye sahip modellerin eğitiminde yüksek paralel hesaplama gereklidir. Araştırmacıların paralel stratejileri nasıl birleştireceklerine, en verimli hızlandırılmış kütüphaneleri seçeceklerine ve FP8 ile FP4 gibi düşük hassasiyet formatlarını entegre edeceklerine karar vermeleri gerekir. Tüm bunları yaparken hız ve bellekten ödün vermemek önemlidir.
Bu süreci kolaylaştıran hızlandırılmış çerçeveler mevcutken, bu özel metodolojilere uyum sağlamanın araştırma ve geliştirme sürecini önemli ölçüde yavaşlattığı görülüyor. Kullanıcılar genellikle tamamen yeni bir kod tabanını öğrenmeleri gerektiğinden, bu durum zaman kaybına neden oluyor.
NVIDIA BioNeMo Tarifleri, büyük ölçekli model eğitimine giriş bariyerini düşürerek süreci basitleştirebilir ve hızlandırabilir. PyTorch ve Hugging Face (HF) gibi tanıdık çerçeveler üzerine inşa edilmiş adım adım kılavuzlar kullanarak, NVIDIA Transformer Motoru (TE) gibi hızlandırılmış kütüphanelerin entegrasyonunun nasıl hız ve bellek verimliliği sağladığını gösteriyoruz. Tamamen Parçalanmış Veri Paralelliği (FSDP) ve Bağlamsal Paralellik gibi tekniklerle performans ölçeklenmektedir.
ESM-2 Modeli ile Hızlandırma Adımları
Bu blog yazısında, Hugging Face ESM-2 protein dil modelini kullanarak, yerel bir PyTorch eğitim döngüsü içerisinde şu adımları gerçekleştireceğiz:
- Transforme Motoru ile hızlandırma.
- Otomatik paralellik için FSDP2 entegrasyonu.
- Daha büyük performans elde etmek için dizilim paketleme gösterimi.
Başlamak için tek gerekenler şunlardır: PyTorch, NVIDIA CUDA 12.8 ve aşağıdaki kaynaklar:
Transformer Motorunun ESM-2 ile Entegrasyonu
TE, transformer hesaplamalarını optimize ederek önemli performans kazançları sağlar, özellikle de NVIDIA GPU’larında. Mevcut eğitim boru hatlarına bütünsel bir revizyon yapmadan entegrasyon sağlamak mümkündür. Bu bölüm, TE’yi ESM-2 gibi bir modele nasıl entegre edeceğimizi BioNeMo tariflerinden ilham alarak gösterecektir.
Çoğu kullanım senaryosunda, TE’nin hazır TransformerLayer modülü kolaylıkla kullanılabilir. Bu, tüm birleşik TE işlemlerini ve en iyi uygulamaları tek bir modülde toplar, böylece gereksiz kod ve kurulum yükü azaltılmış olur. Aşağıda, ESM-2’ye TE entegrasyonunu basitleştiren bir kod kesiti gösterilmiştir. Tam uygulama, bionemo tariflerindeki NVEsmEncoder sınıf tanımında bulunabilir.
import torch
import transformer_engine.pytorch as te
from transformer_engine.common.recipe import Format, DelayedScaling
class MyEsmEncoder(torch.nn.Module):
def __init__(self, num_layers, hidden_size, ffn_hidden_size, num_heads):
super().__init__()
self.layers = torch.nn.ModuleList([
te.TransformerLayer(
hidden_size=hidden_size,
ffn_hidden_size=ffn_hidden_size,
num_attention_heads=num_heads,
layer_type="encoder",
self_attn_mask_type="padding",
attn_input_format="bshd", # veya 'thd', aşağıya bakın.
window_size=(-1, -1), # pencere dikkatini devre dışı bırakın
) for _ in range(num_layers)
])
# İsteğe bağlı olarak gömme, başlık vb. ekleyebilirsiniz.
def forward(self, x, attention_mask=None):
for layer in self.layers:
x = layer(x, attention_mask=attention_mask)
return x
# Katman konfigürasyonu
layer_num = 8
hidden_size = 4096
sequence_length = 2048
batch_size = 4
ffn_hidden_size = 16384
num_attention_heads = 32
dtype = torch.bfloat16
# BSHD formatı için sentetik veri (batch, seq, hidden)
x = torch.rand(batch_size, sequence_length, hidden_size).cuda().to(dtype=dtype)
attention_mask = torch.ones(batch_size, 1, 1, sequence_length, dtype=torch.bool).cuda()
myEsm = MyEsmEncoder(layer_num, hidden_size, ffn_hidden_size, num_attention_heads)
myEsm.to(dtype=dtype).cuda()
fp8_recipe = DelayedScaling(fp8_format=Format.HYBRID)
with te.fp8_autocast(enabled=True, fp8_recipe=fp8_recipe):
y = myEsm(x, attention_mask=attention_mask)
Eğer mimariniz standart bir Transformer bloğundan sapıyorsa, TE yine de katman seviyesinde entegre edilebilir. Temel fikir, standart PyTorch modüllerini (örn. nn.Linear, nn.LayerNorm) TE rakipleri ile değiştirmek ve maksimum performans kazanımları için FP8 oto-dönüşümünü kullanmaktır. TE, Linear, birleşik LayerNormLinear ve dikkat modülleri gibi birkaç alternatif uygulama sağlar. Tam desteklenen modüllerin listesi için TE dokümantasyonuna göz atabilirsiniz.
Verimsiz Dizilim Giriş Verisi Formatları
Standart giriş veri formatları, örneklerin farklı dizilim uzunluklarına sahip olduğunda etkisiz hale gelebilir. Örneğin, ESM-2’nin bağlam uzunluğu 1.024 ile eğitim yapılırken, yaklaşık %60 padding token’ları içerebilir ve bu da modelin dikkat mekanizmasında yer almayan token’lar üzerinde hesaplama kaybına neden olur. Ağlar genellikle giriş dizilerinin gizli durumlarını dört boyutlu bir tensörde temsil eder: [batch boyutu (B), maksimum dizilim uzunluğu (S), dikkate alınan baş sayısı (H) ve başın gizli boyutu (D)], veya BSHD.
Buna alternatif olarak, modern dikkat çekirdekleri, kullanıcıların doldurma token’ları olmadan paketlenmiş girdiler sağlamalarına olanak tanır ve giriş dizileri arasındaki sınırları belirtmek için indeks vektörlerini kullanır. Burada, gizli durumlar, boyutu [düzleştirilmiş girdi token’ları (T), dikkate alınan baş sayısı (H), baş gizli boyutu (D)] olan düzleştirilmiş bir tensör olarak temsil edilir veya THD. Figure 1, bu format değişikliğinin bellek kullanımı ve token verimliliğini artırarak padding token’larını (gri) ortadan kaldırdığına dair bir karşılaştırma sunmaktadır.
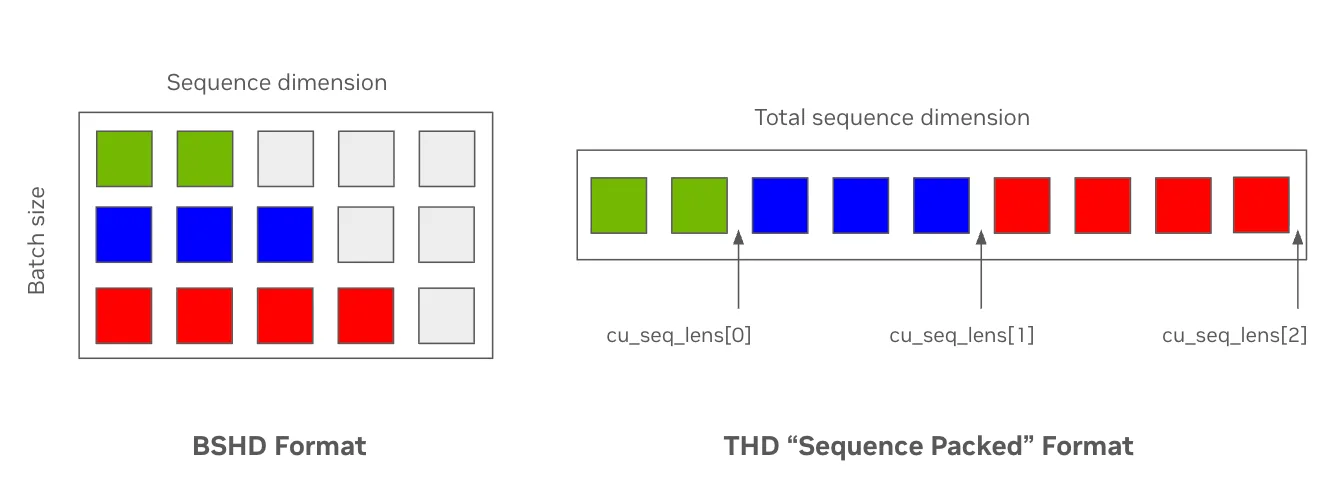
TE, bu optimizasyonu mevcut katmanlara attn_input_format parametresi ekleyerek oldukça basit hale getirir; bu parametre, standart flash-dikkat tarzı kümülatif dizilim uzunluğu anahtar kelime argümanlarını (cu_seq_lens_q) kabul eder. Bu değerler, Hugging Face’in DataCollatorWithFlattening gibi THD’ye uygun toplayıcılar kullanılarak üretilebilir veya BioNeMo Tarifleri’nde uygulanan masking versiyonu gibi farklı yöntemlerle çıkarılabilir.
def sequence_pack(input_ids, labels):
# input_ids dizilerinin listesi: [(S1,), (S2,), ..., (SN,)] şeklinde (B,S)
# Düzleştir ve dizilim sınırlarını takip et
# Her dizinin uzunluğunu belirleyin
sample_lengths = [len(sample) for sample in input_ids]
# input_ids ve etiketleri düzleştir
flat_input_ids = [token for sample in input_ids for token in sample]
flat_labels = [label for sample in labels for label in sample]
# Dizilerin nerede başladığını/stopunu gösteren kümülatif toplamlar oluşturun
# Not: kendi dikkat için cu_seqlens_q ve cu_seqlens_kv aynı olacaktır
cu_seqlens = torch.cumsum(torch.tensor([0] + sample_lengths), dim=0, dtype=torch.int32)
max_length = max(sample_lengths)
return {
"input_ids": torch.tensor(flat_input_ids, dtype=torch.int64),
"labels": torch.tensor(flat_labels, dtype=torch.int64),
# Bu, `flash_attn_varlen_func` gibi yöntemlerce yer alan aynı anahtar kelimelerdir.
"cu_seqlens_q": cu_seqlens,
"cu_seqlens_kv": cu_seqlens,
"max_length_q": max_length,
"max_length_kv": max_length,
}
TE ve Dizilim Paketleme Performansı
Şekil 2’de, TE’nin kullanılması ile birlikte token verimliliğindeki önemli artışı gösteren performans karşılaştırması yer almaktadır. Bu, TE’nin NVIDIA GPU’larınızın hesaplama verimliliğini maksimum seviyeye çıkarmadaki yeteneğini ortaya koymaktadır.
EvolutionaryScale, bir sonraki nesil modellerinde NVIDIA Transformer Motorunu entegre etmiştir:
“ESM3, biyolojik veriler üzerine eğitilmiş en büyük temel modeldir. NVIDIA Transformer Motorunu entegre etmek, 98B parametre ölçeğinde yüksek verimlilik ve GPU kullanım oranıyla eğitmek için kritikaldi,” diyor Tom Sercu, EvolutionaryScale şirketinin kurucu ortağı ve Mühendislik Başkan Yardımcısı. “FP8 hızlandırmanın kesinliği ve hızı, birleşik katmanlar için optimize edilmiş çekirdekler ile birlikte, hesaplama ve model ölçeği sınırlarını zorlamamıza olanak tanıyor. Bu, bilim topluluğu için biyolojiyi anlama yolunda yeni ufuklar açıyor.”
Hugging Face Uyumluluğu
TE’nin önemli avantajlarından biri, Hugging Face gibi mevcut makine öğrenimi ekosistemleri ile uyumluluğudur. Bu, Hugging Face Transformers kütüphanesinden yüklenen modellerle çalışırken de TE’nin performans avantajlarını kullanmanıza olanak tanır.
TE katmanları doğrudan bir Hugging Face Transformers PreTrainedModel içerisinde yer alabilir ve AutoModel.from_pretrained ile tamamen uyumludur. NVIDIA BioNeMo Koleksiyonu üzerinden optimize edilmiş modeller mevcut.
Genellikle, Hugging Face modelinizi yükleyip standart PyTorch katmanlarını (örneğin, nn.Linear, nn.LayerNorm ve nn.MultiheadAttention) TE ile optimize edilmiş karşıtlarıyla değiştirmek için dikkatli bir süreç gerektirmektedir. Bu, bazı katmanları yeniden adlandırmayı veya TE katmanlarının modelin ileri geçişine doğru entegre edilmesini sağlamak için özelleştirilmiş bir model sarmalayıcı kullanmayı gerektirebilir.
Başlayın
BioNeMo Tarifleri ile amacımız, hızlandırma ve ölçeklenmenin, tüm temel model inşa edenler için erişilebilir olmasını sağlamaktır. Daha güçlü ve pratik bir araç seti oluşturmak için sizlerden geri dönüş almak istiyoruz. Tarifleri denemenizi ve GitHub’ımızda bir pull request (katkıda bulunma isteği) veya sorun bildirmenizi teşvik ediyoruz.