Birçok yaşam bilimleri araştırmacısı için birden fazla ilişkili proteinin dizilerini karşılaştırma yeteneği, temel bir görevdir. Bu genellikle çoklu dizi hizalaması (MSA) şeklinde gerçekleşir ve bu hizalamalardan elde edilen evrimsel bilgiler, proteinlerin yapısı, işlevi ve evrimsel geçmişi hakkında önemli içgörüler sağlar.
Şimdi, MMseqs2-GPU ile, evrimsel bilgi edinimi için güncellenmiş bir GPU hızlandırmalı kütüphane ile, protein dizilerinden içgörü almak her zamankinden daha hızlı!
Basit bir dille ifade etmek gerekirse, bir MSA, protein dizilerindeki kalıntıları (veya amino asitleri) temsil eden harflerden oluşmuş büyük bir matristir. Matrisin ilk satırı, analiz için ilgi alanında olan “sorgu” dizisini içerir ve her kalıntı soldan sağa doğru bir sütuna yerleştirilir.
Sonraki satırlar, en benzerden en az benzere doğru, her sütundaki sorgu ile hizalanmış kalıntıları içeren benzer dizileri temsil eder. Eğer bir kalıntı sorgu dizisiyle eşleşmiyorsa, hizalamada bir yer tutucu boşluk eklenir, genellikle “-” ile gösterilir.
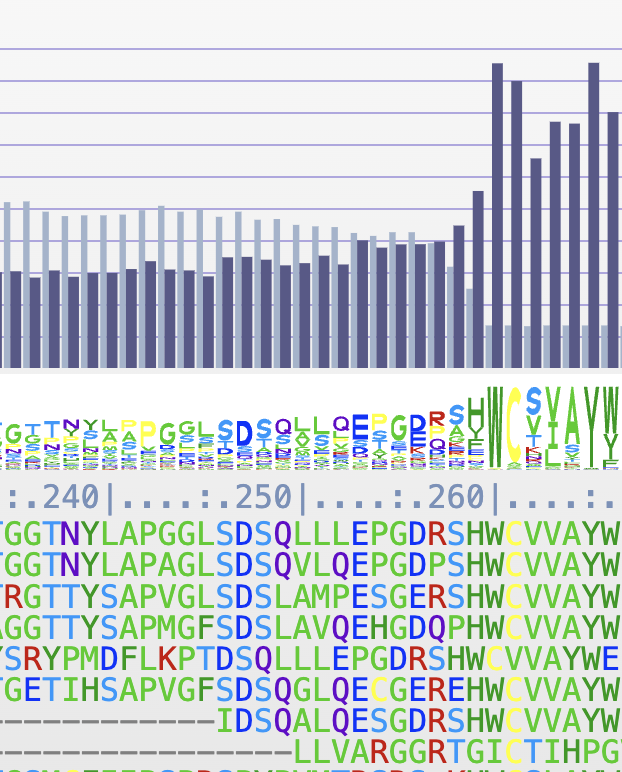
Protein dizilerini içeren büyük bir veritabanından elde edilen MSA’da kodlanmış evrimsel bilgiler, türler arası korunan işlevleri vurgulayan protein alanlarına dair içgörüler sağlar. MSA’daki kalıntıların korunumu üzerine basit bir analiz (yani, bir sütunda aynı amino asidin ne sıklıkla göründüğü), dizideki anahtar kalıntılar hakkında hızlı bir içgörü sunar ki bu kalıntılardaki bir değişiklik, proteinin işlevselliğini etkileyebilir.
Araştırmacılar, inceledikleri proteinler hakkında bilgi geliştirmelerine yardımcı olabilir. MSA’lar, 1992’den beri makine öğrenimi algoritmalarına, karmaşık protein özellikleri gibi yapı ve işlev tahminleri için girdi olarak kullanılmıştır.
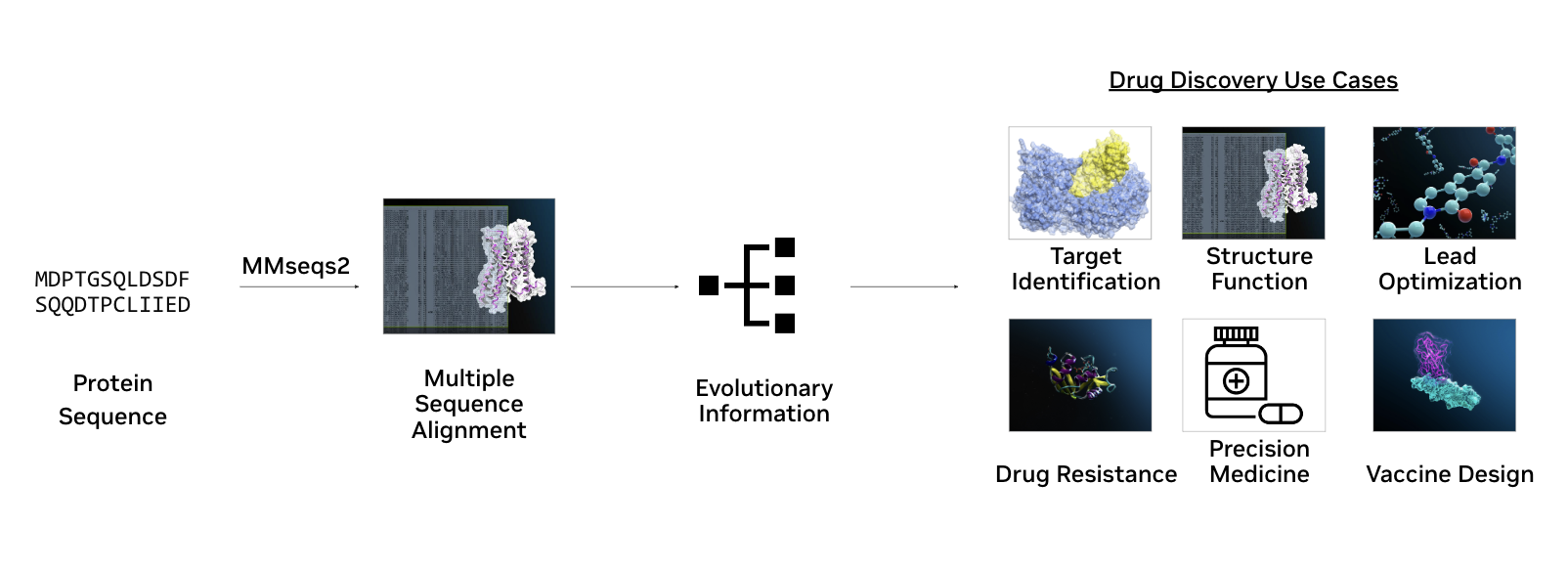
AlphaFold2, hesaplamalı ve yapısal biyolojide devrim yaratmış bir araçtır ve MSA’ları kullanarak son derece doğru 3D protein yapı tahminleri gerçekleştirmektedir; bu, MSA’ların ilaç keşif araştırmalarındaki birçok kullanımdan yalnızca biridir (Şekil 2).
Ancak, MSA hesaplamak zordur, özellikle genel amaçlı CPU’lar, büyük protein dizisi veritabanlarını tarama gibi son derece paralel iş akışları için tasarlanmamıştır. Bu sorun, büyük ölçekli metagenomik deneyler ve ucuz sonraki nesil dizileme sayesinde protein dizisi veritabanları her geçen gün büyüdükçe daha da zorlaşıyor. Bu nedenle, bilgi verici MSA’lar oluşturmak için hızlı bir şekilde büyük veritabanlarını tarayabilen yeni algoritmalar geliştirilmelidir.
NVIDIA CUDA ile Hesaplama Maliyetlerini Aşma
Geleneksel MSA araçları, sırayla işlem yapmada etkili olsa da, GPU’nun paralel işlem yetenekleri ile rekabet edemeyecek olan CPU tabanlı uygulamalara dayanmaktadır.
MMseqs2-GPU’yu geliştiren ortak araştırma ekibine Seoul Ulusal Üniversitesi, Johannes Gutenberg Üniversitesi Mainz ve NVIDIA dâhildir. Önceki CUDASW++4.0 çalışmalarından ilham alan araştırmacılar, bu sorunu, NVIDIA CUDA’ya özel verimli, boşluk içermeyen bir ön filtreleme algoritması geliştirerek ele aldılar. Bu yaklaşım, yüksek hassasiyetle dizileri karşılaştırarak olağanüstü hızda işlem yapılmasını sağlamaktadır.
Bu GPU hızlandırmalı ön filtreleme, MMseqs2’de k-mer ön filtreleme ile değiştirilmiştir. K-mer aramalarını kullanmak yerine, diziler arasındaki karşılaştırmaları basit bir temsil ile sadeleştiren boşluksuz bir puanlama yaklaşımını doğrudan analiz eder. Bu süreç, yalnızca diyagonal bağımlılıkları dikkate alan klasik Smith-Waterman-Gotoh algoritmasının değiştirilmiş bir versiyonunu kullanır; bu sayede hizalamada boşluktan kaçınılmaktadır. Süreç, binlerce GPU çekirdeği üzerinde verimli bir şekilde çalışmaktadır.
Bu algoritmanın, sorgu ile veritabanındaki her dizi arasında çalıştırılmasının sonucu, sorguya en benzer dizileri listeleyen sıralı bir liste olup, bunlar üst düzey adaylar olarak filtrelenebilir; ardından hızlandırılmış affine-gap Smith-Waterman-Gotoh yöntemi uygulanabilir. MMseqs2 kütüphanesine entegre edilen bu algoritmalar, bellek gereksinimlerini azaltmakta ve çoklu GPU sistemleriyle yerel uyumlu hale gelmektedir; bu sayede tek GPU için bellek sıkıntılarını aşmakta ve ek hız artışları sağlamaktadır.
Boşluksuz ön filtreleme adımı, GPU’lar için idealdir çünkü minimum veri aktarımı ile diziden diziye karşılaştırma yapılabilmesine olanak tanır; bu da gecikmeyi azaltır ve GPU kullanımını maksimize eder. Bu yaklaşım sayesinde, tek bir NVIDIA L40S GPU üzerinde çalışan MMseqs2, standart JackHMMER uygulamalarına göre %177 daha hızlı bir performans sergilemiştir (Şekil 3). Sekiz NVIDIA L40 GPU kullanıldığında bu hızlandırma 720 katına çıkar (saniyede 0.117 saniye).
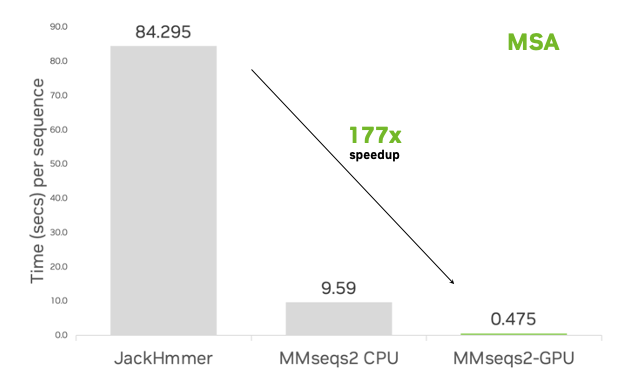
Bu veriler, 30 milyon diziden oluşan bir referans veritabanına karşı hizalanan 6,370 protein dizisi için ortalama çalışma süreleri alınarak elde edilmiştir. Algoritmalar, 128 çekirdekli bir CPU’ya, bir terabayt RAM’e, iki terabayt NVMe depolama alanına ve tek bir NVIDIA L40S GPU’ya sahip bir sistemde çalıştırılmıştır.
Bağlam açısından, bir dizinin hizalanmasını hesaplamak için MMseqs2-GPU kullanmanın süresi (0.475 saniye), insanların bilincini oluşturması (~0.3 ile 0.5 saniye), göz kırpması (~0.3 ile 0.4 saniye) ya da yıldırımın çarpması (~0.2 ile 0.5 saniye) kadar bir zamana eşdeğerdir.
CUDA, MMseqs2-GPU için Optimizasyon ve Hızlandırmayı Nasıl Sağlar?
Bu hızlandırmanın kalbinde CUDA yer almaktadır; bu sayede MMseqs2-GPU, boşluk içeren ve boşluk içermeyen hizalamalar için optimize edilmiş hesaplama çekirdeklerini çalıştırabilir. Bu çekirdekler, çoklu iş parçacığı ve bellek paylaşım özelliklerini kullanarak birçok referans dizisini paralel bir şekilde hizalamaktadır.
MMseqs2-GPU, özellikle en yeni NVIDIA GPU’ları olan NVIDIA L40S GPU ile uyumludur. Boşluksuz ön filtreleme ve boşluk içeren hizalamalar için GPU hızlandırmalı çekirdekler, GPU’ların yüksek paralelliğinden yararlanır. Boşluksuz ön filtre, her matris satırını paralel olarak işleyerek, erişimleri optimize etmek için GPU paylaşımlı belleği kullanır ve maksimum verimlilik sağlamak üzere 16-bit sayıları 32-bit kelime içinde paketler (half2 veya s16x2 veri tipleri kullanarak).
Ayrıca, dinamik programlama bağımlılıklarını warp seviyesinde cross-thread warp shuffle kullanarak etkin bir şekilde yönetir. Gerekli bellek erişimleri, hızlı CUDA paylaşımlı belleği kullanılarak hızlandırılır. Bu tekniklerin birleşimi, sorunu hesaplama odaklı hale getirir ve bellek erişimlerinden kaynaklanan fazla yükü en aza indirir.
Araç, ölçeklenebilirliği sağlamak için çoklu GPU kurulumlarını destekler; böylece araştırmacılar, birden fazla GPU arasında hesaplama yüklerini dağıtarak daha büyük veri setlerini işleyebilir. Bu mimari, bulut tabanlı ortamlara oldukça uyum sağlayarak, araştırmacıların maliyetleri düşürmeden kaydedilen doğruluklarından ödün vermeden büyük projeleri yönetmelerine olanak tanır.
“Böyle bir şeyi uzun zamandır bekliyorduk. Protein yapı tahmini çıkarımı uzun süredir MSA hesaplama aşaması ile sınırlı olduğunu biliyoruz. Bu, inanılmaz bir başarı; MSA adımını %20’den daha az bir süreye indirgemek, yapı tahmin iş akışlarına yaklaşma şeklimizi tamamen değiştiriyor,” diyor VantAI’daki CTO Luca Naef.
MMseqs2-GPU, Protein Yapı Tahminini Hızlandırıyor
MMseqs2-GPU’nun başarısı, boşluk içermeyen ön filtreleme ve boşluk içeren hizalama algoritmalarını yeniden tasarlamakta ve CUDA’yı kullanarak hızlı, uygun maliyetli ve ölçeklenebilir bir dizilim sunmakta yatmaktadır; bu, günümüzün biyoistatistik araştırma taleplerini karşılamaktadır.
MMseqs2, GPU kullanılarak birçok hesaplama sürecinde, Colabfold ile yapı tahmini gibi, kullanıcılarına kolayca takviye edilmiş bir performans artışı sunmaktadır:
Hız Artışı
Colabfold, MMseqs2-GPU kullanarak, JackHMMER ve HHblits ile AlphaFold2’ye göre 22 kat daha hızlıdır (Şekil 4). Pratikte bu, HHblits, JackHMMER ve AlphaFold2 kullanarak bir protein yapısını tahmin etmek için 40 dakika beklemek yerine, Colabfold ve MMseqs2-GPU kullanarak bu tahmini bir buçuk dakikada alabileceğiniz anlamına gelir.
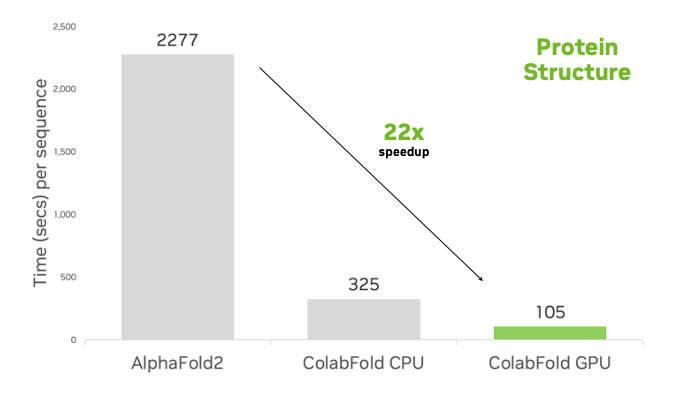
Grafik, 20 CASP14 sorgusu için yapılan tahminlere dayanmakta ve her yöntemin doğruluğu (LDDT) yaklaşık 0.76’tır. Tahmin yöntemleri, 128 çekirdekli bir CPU’ya, bir terabayt RAM’e, iki terabayt NVMe depolama alanına ve tek bir NVIDIA L40S GPU’ya sahip bir sistemde çalıştırılmıştır.
Bellek Gereksinimleri
Boşluksuz GPU ön filtre ile, MMseqs2-GPU, CPU uygulamasında gereken büyük k-mer hash tablolarını kullanmaktan kaçınarak, bu çözümü GPU’lara daha uygun hale getirir ve genel bellek gereksinimlerini bir sıra azaltır (Şekil 3 ve 4 açıklamalarını inceleyin).
Maliyet Etkinliği
Colabfold, MMseqs2-GPU kullanarak, JackHMMER ve HHblits ile AlphaFold2’ye göre bulut maliyet tahminlerinde 70 kat daha ucuzdur. Bu büyük maliyet azalması, özellikle kısıtlı bütçelere sahip laboratuvarlar için güçlü biyoistatistik araçlarına erişimi kolaylaştırır. Düşük hesaplama maliyetleri ayrıca, finansal olarak zahmetli olabilecek devam eden ve büyük ölçekli analizlerin yapılmasına da olanak tanır.
Yüksek Verim ve Ölçeklenebilirlik
Yeni geliştirilen boşluk içermeyen ön filtre, sekiz GPU’da 102 Tera Hücre Güncellemeleri Saniye (TCUPS) hızına ulaşabilir ve büyük veri setlerini hızla ön filtreleyebilir. Araç, çoklu GPU işlemesine destek vererek, kullanıcıların daha büyük veri setlerini işleyebilmelerini ve toplam yürütme hızını artırmalarını sağlamakta; bu, büyük genomik veya proteomik çalışmalarda kritik öneme sahiptir.
Doğruluk
MMseqs2-GPU, bu hız ve maliyet avantajlarını doğrulukta bir kayıp yaşamadan sunmaktadır. Duyarlılığını ve protein katlama doğruluğunu koruyarak, araştırmacıların hızlı içgörüler elde etmesine olanak tanımaktadır.
“Columbia’daki laboratuvarım, OpenFold adıyla AlphaFold2’nin sadık bir reprodüksiyonunu geliştirdi; bu sayede topllu olarak topluluk kendi protein yapı tahmin modellerini eğitebiliyor. Uygulamalarımız için özellikle ilginç olan, daha bilgilendirici MSA’lar sağlamakta çok sayıda iteratif profil arama yapabilme yeteneği. MMseqs2-GPU’nın önceki yöntemlere göre daha hızlı profil aramalarını desteklemesi bizi oldukça heyecanlandırıyor,” şeklinde belirtti Columbia Üniversitesi’nden profesör Mohammed AlQuraishi.
Hızlandırılmış MMseqs2, Daha Hızlı Keşifler Anlamına Geliyor
Gelecekte, ortak araştırma ekibi algoritmaları ve MMseqs2 entegrasyonunu daha da refine etme, protein kümelenmesi ve kademeli veritabanı aramaları gibi uygulamaları genişletme üzerine yoğunlaşmayı planlıyor. MMseqs2’nin sağladığı hızlı girdi, daha hızlı ilaç keşfi sürecine katkı sağlayabilir; bunu burada gösterdik ve diğer birçok uygulamanın yanı sıra (Şekil 2) etkisini görmekteyiz.
Örneğin, GEMME gibi protein varyant tahminlerine daha hızlı girdi sağlayabilir; bu sayede hastalık varyantları hakkında daha derin bir anlayış geliştirebiliriz ve PoET gibi protein LLM için gerçek zamanlı geri getirme sağlanabilir. Ayrıca, daha hızlı antimikor ilaç direnç profilleri oluşturulabilir; hatta daha hızlı aşı tasarımı yapılabilir.
Bu alanda derinlemesine bilgi edinmek veya katkıda bulunmak isteyenler için MMseqs2-GPU açık kaynaklı olup çevrimiçi olarak mevcut. Araştırmacılar için paha biçilmez bir kaynak sunmaktadır.
Daha fazla bilgi için lütfen MMseqs2 GitHub sayfasını ziyaret edin ya da detaylandırılmış analiz ve karşılaştırmalara göz atın. Ayrıca, MMseqs2 ile MSA adımı olarak test edebileceğiniz bir AlphaFold2 NVIDIA NIM de bulunmaktadır.